Para quem só sabe usar martelo, todo problema é um prego. Até rasters em formato netCDF.

Nas últimas semanas estive trabalhando em um projeto com dados atmosféricos. Tem sido de grande aprendizado não apenas por ser uma temática nova, mas também pela demanda de processamento de dados.
Os aprendizados relacionados ao processamento de dados (sua otimização, claro), serão abordados em artigos ainda em produção. Mas um ponto fundamental e que percebi ser ignorado pela maioria, e neste sentido me incluo, é a estrutura de dados dos arquivos raster em netCDF.
Já vi, e já respodi a muitas pessoas perguntando como carregar dados netCDF no R, com um simples “com o pacote raster, ora bolas”. No pior das hipóteses, teremos apenas que instalar uma dependencia que é o pacote ncdf4.
Pois é, os dados atmosféricos que tenho trabalhado estão em formato netCDF. E usar o pacote raster para carregá-lo e manipulá-lo, me foi útil enquanto a análise era exploratória. Ao ver o volume de dados crescer de três dias para dois meses e, em seguida para dois anos de dados produzidos a cada três horas do dia, todos os dias, totalizando 731 arquivos, percebi que precisaria deixar de preguiça e entender esse tal de netCDF.
Lendo sobre o pacote ncdf41, o formato netCDF é descrito da seguinte forma:
“binary data files that are portable across platforms and include metadata information in addition to the data sets.”
Acho nesse fragmento já dá para entender a importância do formato, não?
Vamos entender isso? Vejam a diferença:
library(raster)
## Loading required package: sp
(r.ppm25 <- stack("./rasters/ppm25_2019-06-30.nc"))
## Loading required namespace: ncdf4
## class : RasterStack
## dimensions : 59, 75, 4425, 6 (nrow, ncol, ncell, nlayers)
## resolution : 0.4, 0.4 (x, y)
## extent : -74.19, -44.19, -18.24, 5.36 (xmin, xmax, ymin, ymax)
## crs : +proj=longlat +datum=WGS84 +ellps=WGS84 +towgs84=0,0,0
## names : X2019.06.30.12.00.00, X2019.06.30.15.00.00, X2019.06.30.18.00.00, X2019.06.30.21.00.00, X2019.07.01.00.00.00, X2019.07.01.03.00.00
Aos que já trabalham com o pacote raster e conhecem os dados raster, poderão ver que, sim, ainda que dependendo do ncdf4, o raster carrega os dados, aparentemente, sem grandes problemas. O mínimo esperado está aí. Um raster, com um extent, linhas, colunas, células, layers…
Mas, se os dados estão em netCDF, e esse tipo de dados permite armazenar metadados também, cadê eles?
Pois é. Ao ignorar isso, passava por cima desse detalhe como um marreteiro diante de um parafuso.
Vamos abrir o arquivo com o ncdf4 para começar a identificar alguns pontos importantes, que, por sinal não ficam “apenas” no metadados:
library(ncdf4)
(nc.ppm25 <- nc_open("./rasters/ppm25_2019-06-30.nc"))
## File ./rasters/ppm25_2019-06-30.nc (NC_FORMAT_64BIT):
##
## 1 variables (excluding dimension variables):
## short pm2p5[longitude,latitude,time]
## scale_factor: 9.668789576322e-12
## add_offset: 3.16807559257767e-07
## _FillValue: -32767
## missing_value: -32767
## units: kg m**-3
## long_name: Particulate matter d < 2.5 um
## standard_name: mass_concentration_of_pm2p5_ambient_aerosol_particles_in_air
##
## 3 dimensions:
## longitude Size:75
## units: degrees_east
## long_name: longitude
## latitude Size:59
## units: degrees_north
## long_name: latitude
## time Size:6 *** is unlimited ***
## units: hours since 1900-01-01 00:00:00.0
## long_name: time
## calendar: gregorian
##
## 2 global attributes:
## Conventions: CF-1.6
## history: 2021-03-17 20:27:56 GMT by grib_to_netcdf-2.20.0: grib_to_netcdf /data/scratch/20210317-2020/8e/_mars-webmars-public-svc-blue-001-6fe5cac1a363ec1525f54343b6cc9fd8-ZAB6WD.grib -o /data/scratch/20210317-2020/b1/_grib2netcdf-webmars-public-svc-blue-000-6fe5cac1a363ec1525f54343b6cc9fd8-VDR6DW.nc -utime
Antes de comentar toda riqueza e detalhamento de informação que nos brinda o ncdf4, percebam que o comando é nc_open(). Não importa de o dado tem uma ou mais “bandas” ou “camadas”. Isso me chamou atenção pois, como acham que vou saber a quantidade de camadas do dados que vou abrir, se ainda não o conheço? Mas enfim. Vamos ao mais importante, os detalhes:
Veja a estruturação. No arquivo em questão, eu tenho uma variável apenas, que possui vários metadados, garantindo minimamente sua compreensão, como: short_name, no caso “pm2p5”, units, para descrever a unidade do dado, long_name e standart_name, além de vários outros.
E vejam também que são identificadas três dimensões: longitude, latitude e time;
Eis que chegamos ao cubo de dados, não faz sentido?
Eu tenho uma variável (ppm<2.5) medida para uma mesma área (cena) ao longo do tempo. O netCDF, como está preparado e pensado para esse tipo de situação já entende os dados como informação de dadta e hora (!) e os organiza como uma dimensão do dado e não atributo. O raster, por ter outros objetivos, carrega tudo como um stack (empilhamento de camadas) e adiciona uma string “X” ao nome das camadas, já que as mesmas são numéricas e o pacote em questão restringe o prefixo das camadas a texto, não permitindo valores numéricos. Enfim, nos obriga a digitar mais algumas linhas de código para que o dado fique mais apresentável.
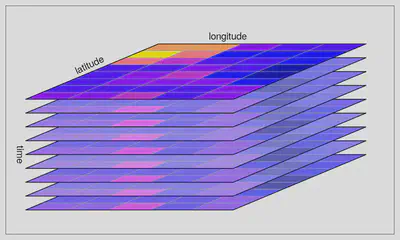
Fonte da imagem: r-spatial.org
Essa forma de estruturar os dados fará todo o sentido mais à frente quando formos falar um pouco mais do processamento desses dados.
O pacote ncdf4 está desenvolvido para o que se convêm chamar de CRUD, na área de desenvolvimento de sistemas: Create, Update e Delete. Ou, seja, a ideia é fornecer ferramentas para manipular e ter controle total dos dados, criando, abrindo, alterando dimensões ou atributos e por aí vai. Mas não para visualização nem para as análises. Essas, ficarão para os próximos artigos.
Enquanto isso, fique à vontade em me contactar, dar uma alô lá no grupo do GeoCastBrasil, no Telegram ou no nosso canal do youtube.
Não pretendo explorar muitos detalhes, mas o nome do pacote (
ncdf4) faz referência à versão 4 de estratura em questão. Portanto, caso você esteja, por algum motivo, usando um arquivo netCDF com versão anterior, o pacotencdfsegue disponível, ainda que descontinuado. ↩︎